さまざまな薬の合成に重要な酵素が作用する相手を認識する仕組み キラルアミン合成への酵素利用の拡大

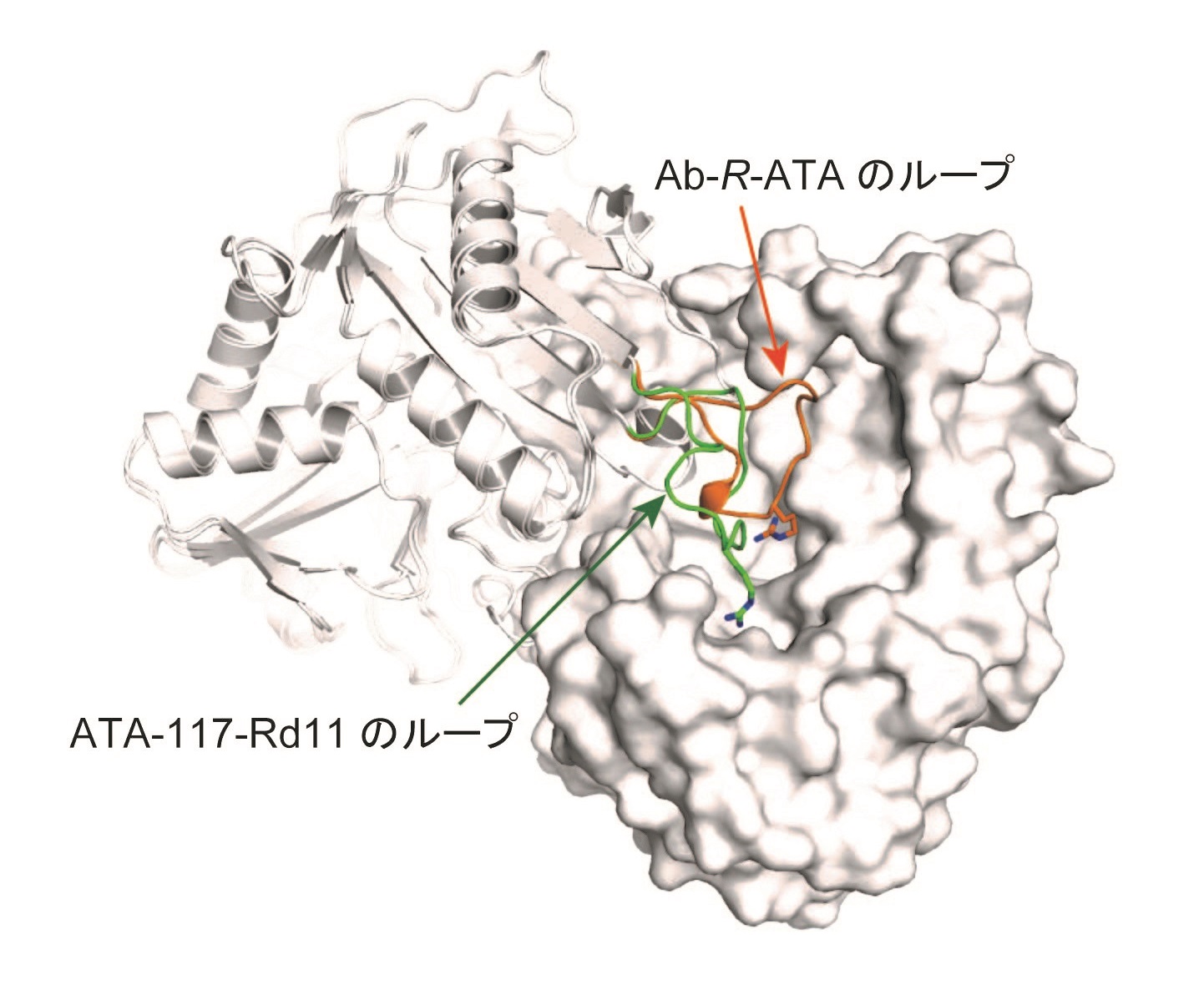
Ab-R-ATAと人工的に合成したATA-117-Rd11のループ構造の比較
Ab-R-ATAとATA-117-Rd11を重ね合わせて各々の二量体構造を表示している。二量体を形成するプロトマーの一方は分子表面を表示する方法で示しており(右)、もう一方はリボン図(左)で示している。Ab-R-ATAとATA-117-Rd11の活性部位に隣接するループ構造は、それぞれオレンジと緑で示し、構造の違いが明瞭である。
© 2015 田之倉 優
東京大学大学院農学生命科学研究科の田之倉優教授らの研究グループは、2型糖尿病の薬を工業的に合成する過程で用いられている酵素の結晶構造を明らかにし、その酵素が基質を認識するメカニズムを解明しました。この成果は、産業上有用なキラルアミン合成に活用されることが期待されます。
キラルアミンは、医薬、農薬、他の産業上有用な化合物の合成に欠かせない構成要素です。高効率なキラルアミン生産を実現するためのツールとして、アミントランスアミナーゼ(ATA)が注目されてきました。特にR体のキラルアミンを合成できるATA(R-ATA)は利用価値が高く、2010年に米国の製薬会社は2型糖尿病に対する経口血糖降下薬sitagliptinの合成に適用できるよう既存のR-ATAを酵素改変することに成功しました。これにより、従来の化学触媒法と比べて、一日当たりの生産力を53%増加させ、さらに19%の製造コストの削減を達成しています。
その一方で、これほど工業的に重要な酵素のR-ATAがどのようにして基質を認識しているかなど、まだ明らかになっていない点が残されていました。
今回、田之倉優教授らの研究グループは、株式会社カネカ バイオテクノロジー開発研究所等との共同研究により、土壌などに生息する細菌(Arthrobacter sp. KNK168)に由来する R-ATA(Ab-R-ATA)の立体構造をX線結晶解析により決定し、R-ATAが有する基質認識のメカニズムとそのメカニズムを決定する構造基盤を解明しました。Ab-R-ATAは同一の活性部位で2種類の基質を認識しますが、活性部位にあるアルギニン(Arg138)は両方の基質認識に機能していました。さらに、人工的に合成したR-ATAとAb-R-ATA に変異を加えた(G136F;136番目のグリシンをフェニルアラニンに置換)酵素の結晶構造の解析により、R-ATAの活性部位に隣接するループ構造が基質特異性改変の標的部位であることが示されました。
「本研究で得られた知見は、多様なキラルアミン化合物を創製するための新しい触媒の開発を加速し、R-ATAを触媒として用いたキラルアミン合成の産業応用への貢献が期待されます」と田之倉教授は話します。
論文情報
, "A new target region for changing the substrate specificity of amine transaminases", Scientific Reports: 2015/06/01 (Japan time), doi:10.1038/srep10753.
論文へのリンク(掲載誌、UTokyo Repository)