原始的な代謝で働く単純な酸化還元酵素のかたち 古い酵素から新しい薬への展望

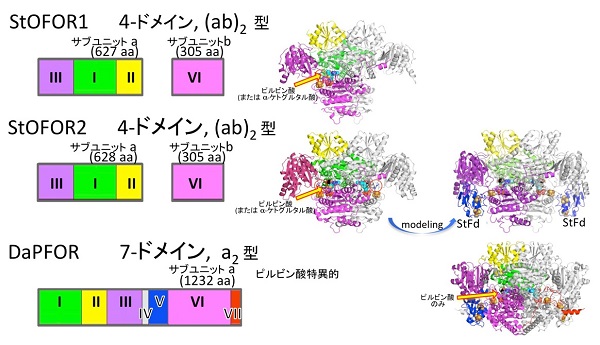
原始的な代謝系で働く単純な酸化還元酵素のドメイン構成と立体構造
超高温で生きる古細菌から得られた単純なドメイン構成の酵素(StOFOR1)が複雑な構成の酵素(DaPFOR)よりも多様な基質特異性を示す理由が、立体構造から明らかになりました。
© 2016 Takayoshi Wakagi.
東京大学大学院農学生命科学研究科の若木高善前教授らの研究グループは、原始的な細胞内の代謝系で働く2-オキソ酸:フェレドキシン酸化還元酵素(OFOR)の中で、最も単純な構造でありながら多様な分子の反応を促進する(基質特異性の広い)酵素を、高い温度でも生存できる超好熱性古細菌スルホロバスから取得して、その立体構造を初めて決定しました。その構造・機能との相関が明らかになったことで、今後、生命の起源に関わる原始的な代謝系の理解や、OFORを有する病原性微生物を駆除するという応用につながると期待されます。
研究グループがスルホロバスの細胞から最初に取り出したOFORのうち、酵素StOFOR1と呼ばれる酵素は、ピルビン酸やケトグルタル酸などの反応を広く促進できることがわかっていました。しかし、唯一立体構造が報告されていた細菌由来のピルビン酸専用のOFOR(DaPFOR)が7個のドメイン(特定の機能をもったタンパク質内の一部の構造)から構成されているのに対し、StOFOR1は4個のドメインしかありません。それにもかかわらず、基質特異性が広く、中央代謝やアミノ酸代謝で重要な働きをする、その構造的な基盤は不明でした。
酵素の機能を理解するためには、その精密な立体構造を決めることが重要です。研究グループは、StOFOR1とその類似酵素であるStOFOR2の3次元的な構造を、X線結晶構造解析法を用いて明らかにしました。これら2つの酵素とDaPFORを比べると、ドメインの配置順序が異なるにもかかわらず、全体的な立体構造の中心部分は類似していました。さらに構造が既に知られているスルホロバス細胞のフェレドキシンとStOFORとを組み合わせて、酸化反応を完了することが可能な構造を作ると、いっそうDaPFORに類似していました。しかし、StOFORの2-オキソ酸結合部位はDaPFORと比べると、ピルビン酸に限らずケトグルタル酸も結合できるだけの奥行きがあり、結合にかかわるアミノ酸配列も変異体解析法によって確認しました。
「20年間取り組んできた2-オキソ酸:フェレドキシン酸化還元酵素の構造がようやく解析できました」と若木前教授は話します。「今後、ドメイン配置の異なるものや病原微生物の2-オキソ酸:フェレドキシン酸化還元酵素の構造解析が進めば創薬にもつながるでしょう」と期待を寄せます。
論文情報
, "Crystal structures of archaeal 2-oxoacid:ferredoxin oxidoreductases from Sulfolobus tokodaii", Scientific Reports Online Edition: 2016/09/13 (Japan time), doi:10.1038/srep33061.
論文へのリンク(掲載誌)