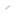新規セリン生合成酵素の生物間分布 セリン合成酵素として働くのに必須な構造要因の特定

東京大学農学生命科学研究科・応用微生物学研究室および食品生物構造学研究室のグループは新規タンパク質のX線結晶構造解析を行い、その機能に重要な因子を特定した。これにより、様々な生物におけるセリン生合成経路を知ることが可能になった。
これまでに応用微生物学研究室の研究グループは、「絶対独立栄養性水素細菌Hydrogenobacter thermophilus」という細菌が、既知の合成酵素ホスホセリン脱リン酸化酵素(PSP)とは起源が全く異なる新規の酵素を用いて、アミノ酸の一種であるセリンを合成していることを突き止めていた。これはセリン生合成の新規経路の発見であった。そして、この新規PSPに類似のタンパク質が多種多様な生物に存在し、セリンの生合成に寄与していることも予測していた。しかし、この新規PSPに類似のタンパク質の基質選択性は非常に複雑であり、遺伝子データベース上の新規PSP類似タンパク質遺伝子が本当にPSPとして機能しているのか、それとも他の機能を有するのか見分けるのは困難であった。そこで応用微生物学研究室および食品生物構造学研究室のグループは新規PSPのX線結晶構造解析を行い、PSPとして機能するのに重要な因子を特定した。
新規PSPの高分解能結晶構造から、ホスホセリンと相互作用可能な85番目のヒスチジン側鎖および活性ポケットの蓋となるC末端のアミノ酸数残基が新規PSPの特徴であることが示唆された。これらの部位に変異を加えたタンパク質を解析した結果、これらの部位がPSPとして機能するのに非常に重要であることが実証された。さらに、他生物が有する新規PSPに類似のタンパク質を解析した結果、上述の2つの因子を有するもののみが高いPSP活性を有し、セリン生合成酵素として機能可能なことが明らかになった。
以上の結果から、新規PSPと同様にPSPとして働いているタンパク質をゲノムデータベースから高精度で予測することが可能になった。これにより、絶対独立栄養性水素細菌だけでなく、シアノバクテリアなどの性質の異なる生物にも新規PSPが存在することが明らかになり、様々な生物におけるセリン生合成経路を知ることができた。アミノ酸合成という、生物にとって非常に基本的な機能に多様性があることを示した本成果は、生物の進化の解明に非常に重要であると言える。
論文情報
Yoko Chiba, Shoichiro Horita, Jun Ohtsuka, Hiroyuki Arai, Koji Nagata, Yasuo Igarashi, Masaru Tanokura, Masaharu Ishii,
“Structural Units Important for Activity of a Novel-type Phosphoserine Phosphatase from Hydrogenobacter thermophilus TK-6 Revealed by Crystal Structure Analysis”,
The Journal of Biological Chemistry 288 (2013):11448-11458, doi: 10.1074/jbc.M112.449561.
論文へのリンク