約24時間周期の体内時計が生み出される仕組み サーカディアンリズムの鍵分子が結合するゲノム領域とDNA塩基配列を決定

睡眠と覚醒サイクルやホルモンリズムなど多くの生理現象は約24時間周期のリズムを刻んでいます。このような生理現象の周期性をサーカディアンリズムと呼び、サーカディアンリズムは、転写や翻訳を介した負のフィードバックループにより自律的に制御されています。分子レベルでは、一定のリズムでDNAに結合する分子(CLOCK-BMAL1転写因子複合体)が、さまざまな遺伝子の発現を制御し、サーカディアンリズムの制御において中心的な役割を担っていることが知られていました。しかし、分子レベルにおけるサーカディアンリズムの制御についての詳細は未だ不明な点が多く残されています。
© 2014 吉種 光
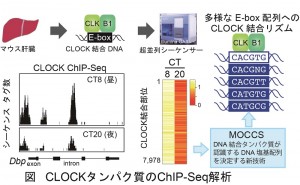
マウス肝臓を用いたChIP-Seq解析によりCLOCKタンパク質のDNA結合を調べた。Dbp伝子周辺のシーケンスタグ数のプロファイルやヒートマップ解析の結果、CLOCKは一定のリズムでDNAに結合していることが分かる。このChIP-SeqデータをもとにMOCCS解析を行い、CLOCKが結合するDNA領域を決定した。
東京大学大学院理学系研究科の吉種光助教、寺嶋秀騎大学院生、深田吉孝教授らの研究グループは、新領域創成科学研究科の尾崎遼大学院生および大気海洋研究所の岩崎渉准教授の研究グループらと共同で、マウスにおいて転写因子CLOCKが実際に結合しているDNA領域を、次世代シークエンサーを用いて網羅的に同定しました。また、サーカディアンリズムは、転写因子CLOCKがDNAに結合する直接的な制御に加えて、他の転写因子や遺伝子発現を抑制する短いRNA配列(マイクロRNA)による間接的な制御により巧みに調節されていることが分かりました。さらに、研究グループは、新しいバイオインフォマティクス手法MOCCS(motif centrality analysis of ChIP-Seq; ChIP-Seq解析で得られたデータを利用し、DNA結合タンパク質が認識するDNAの塩基配列を決定する技術)を開発し、転写因子CLOCKが認識するDNAの塩基配列を決定することに成功しました。
MOCCSはサーカディアンリズムに限らず、DNAに結合するさまざまなタンパク質について、実際に結合するDNA領域を網羅的に決定しようとする研究に応用されることが期待されます。
論文情報
Hikari Yoshitane, Haruka Ozaki, Hideki Terajima, Ngoc-Hien Du, Yutaka Suzuki, Taihei Fujimori, Naoki Kosaka, Shigeki Shimba, Sumio Sugano, Toshihisa Takagi, Wataru Iwasaki, and Yoshitaka Fukada,
“CLOCK-controlled polyphonic regulations of circadian rhythms through canonical and non-canonical E-boxes”,
Molecular and Cellular Biology 2014, May, 34(10), 1776-1787, doi: 10.1128/MCB.01465-13.
論文へのリンク