RNAとタンパク質の両方をリン酸化する酵素を世界で初めて発見 ―遺伝暗号の解読に必須な修飾塩基アグマチジンの生合成を解明―研究成果

「RNAとタンパク質の両方をリン酸化する酵素を世界で初めて発見 |
平成23年10月17日
東京大学大学院工学系研究科
1.発表者:
鈴木 勉(東京大学大学院工学系研究科化学生命工学専攻・教授)
木村 聡(同・大学院生、日本学術振興会 特別研究員)
寺坂尚紘(同・大学院生)
沼田倫征(産業技術総合研究所バイオメディカル研究部門RNAプロセシング研究グループ・研究員)
大澤拓生(同・協力研究員、日本学術振興会 特別研究員)
2.発表概要:
DNAに書かれた遺伝暗号を精確に読み取ることは、すべての生命に課せられたもっとも根本的で重要なタスクです。アグマチジン(agm2C)は、アーキア(古細菌)の転移RNA(tRNA) 注1)に存在する修飾塩基であり、遺伝暗号の解読においてAUAコドン注2)の認識に必須であることが知られています。東京大学大学院工学系研究科の鈴木 勉教授の研究グループは、産業技術総合研究所バイオメディカル研究部門の沼田倫征研究員のグループと共同で、この修飾塩基の生合成酵素であるアグマチジン合成酵素TiaS(tRNAIle-agmatidine synthetase)を同定し、組換えタンパク質を用いて試験管内でのアグマチジン再構成に成功しました。詳細な反応機構の解析およびTiaS-tRNA複合体のX線結晶構造解析の結果、TiaSが、RNAとタンパク質の両方の基質をリン酸化するこれまで知られていなかった新規キナーゼドメイン(TCKドメインと命名)を有していることが明らかとなり、全く新しい酵素触媒反応によってアグマチジンが形成される機構を解明しました。この研究は、生体内の化学反応の理解に貢献しただけでなく、遺伝暗号解読機構の成立基盤を解明した重要な成果といえます。
3.発表内容:
あらゆる生物はDNAに書かれた遺伝情報を基にしてタンパク質を合成しています。4種類の塩基が3つ組で1つのコドンとなり、アミノ酸を指定するという考え方は、半世紀前に確立され、セントラルドクマ(中心教義)として、生命科学の根幹を貫く最もエレガントで美しい原理の一つです。ところが、64通りの遺伝暗号がどのようなしくみで正確に解読されるかは、古典的なテーマであるものの、いまだ完全には解明されていないのが現状です。
アーキア(古細菌)において、AUAコドンをどのようにアミノ酸であるイソロイシンへと解読するかについては、長年、この分野の研究者にとっての大きな課題でした。昨年、鈴木教授のグループは、アーキア由来tRNAIleのアンチコドン注3)から新規の修飾塩基であるアグマチジン(agm2C)を発見しました(図1)(Ikeuchi et al., Nature Chem Biol., 6, 277-282, 2010)。
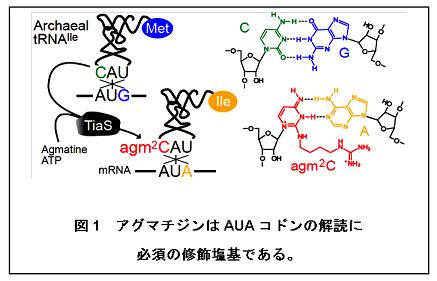
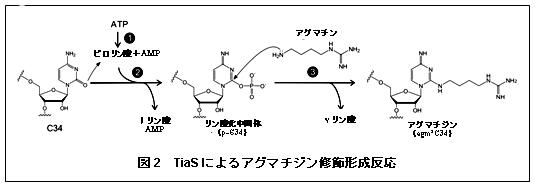
アグマチジンは、シチジン(C)の2位にアグマチン(アミノ酸の一種であるアルギニンから二酸化炭素が脱落したもの)が結合したものであり、この修飾によってシトシン環が互変異性化注4)し、グアニン(G)とは対合できずアデニン(A)と対合することで、AUAコドンを解読する機構が明らかとなっています(図1)。また、アグマチジンの生合成酵素については、比較ゲノム注5)の手法を用いて同定され、TiaS(tRNAIle-agmatidine synthetase)と命名されました。TiaSはアグマチンとATPを基質として、tRNAIleのアンチコドン1字目のシチジン(C34)を修飾し、アグマチジンを形成する酵素です。今回の研究で、アグマチジンの生合成機構が詳細に調べられたところ、TiaSは三段階の酵素反応を触媒することが明らかとなりました(図2)。まず、ATPをAMPとピロリン酸に加水分解します。次に、C34の2位のカルボニル基をリン酸化することにより活性化します。最後にアグマチンのアミノ基にC34の2位の炭素原子を求核置換攻撃させることで、リン酸基が脱離し、アグマチジンが形成されます(図2)。この触媒反応で驚くべきことは、TiaSがtRNAをリン酸化する前に、ATPをAMPとピロリン酸に加水分解する点です。このような反応を触媒する酵素(ATP加水分解酵素やキナーゼ)はこれまでに知られておりません。
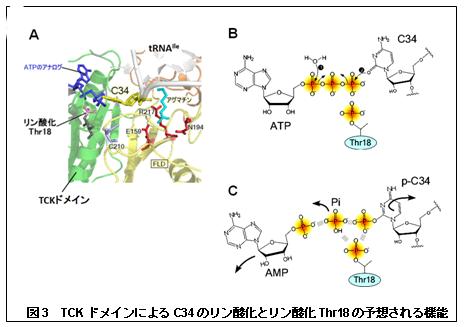
さらに、TiaSは自身の18位にある保存されたトレオニン残基(Thr18)をリン酸化することを見出しました(図3)。リン酸化されたThr18はagm2C形成反応に重要な役割を担っていると考えられています。共同研究先である産業技術総合研究所の沼田研究員のグループは、TiaS-tRNAIle複合体の結晶構造を解明し(Osawa et al., Nature Struct & Mol Biol, in press)(図3A)、TiaSは、RNA(C34)とタンパク質(Thr18)の異なる基質をリン酸化する、これまで知られていなかった新規ドメインを有することが明らかとなりました。このドメインはTCK(Thr18-C34 kinase)ドメインと命名されました。リン酸化Thr18はTCKドメインのATP結合部位に存在し、特にβとγリン酸基の近傍に位置しています。したがって、リン酸化Thr18とγリン酸基の間には負電荷同士の反発が生じ、ピロリン酸からC34へのリン酸化を促進している可能性があります(図3B)。さらにリン酸化後に、リン酸化Thr18とリン酸化C34の間で負電荷同士の反発が生じ、リン酸化C34をアグマチン結合ポケットまで移動させる役割(図3C)があるのではないかと推察されます。
これまでに、RNAのみ、タンパク質のみをリン酸化する酵素はたくさん知られてきましたが、本研究でRNAとタンパク質という全く異なる生体高分子をリン酸化する酵素が世界で初めて見つかりました。この成果は、RNA分野のみならず、タンパク質科学や酵素学など生命科学全般において大きなインパクトを与えるものであります。また、遺伝暗号の成立基盤を分子レベルで明らかにした成果でもあります。
4.発表雑誌および問い合わせ先:
雑誌名:Nature Structural & Molecular Biology (オンライン版の場合: 10月16日)
論文タイトル:Biogenesis of 2-agmatinylcytidine catalyzed by the dual protein and RNA kinase TiaS
著者: Naohiro Terasaka, Satoshi Kimura, Takuo Osawa, Tomoyuki Numata & Tsutomu Suzuki
問い合わせ先:東京大学大学院工学系研究科 化学生命工学専攻 教授 鈴木 勉
雑誌名:Nature Structural & Molecular Biology (オンライン版の場合: 10月16日)
論文タイトル:Structural basis of tRNA agmatinylation essential for AUA codon decoding
著者: Takuo Osawa, Satoshi Kimura, Naohiro Terasaka, Hideko Inanaga, Tsutomu Suzuki & Tomoyuki Numata
問い合わせ先:産業技術総合研究所バイオメディカル研究部門RNAプロセシング研究グループ 研究員 沼田倫征
5.用語解説:
注1)転移RNA (tRNA)
遺伝暗号(コドン)を解読するアダプター分子で、約75塩基長のRNA。コドン(注2)に対応するアミノ酸種ごとにそれぞれのtRNAが存在する。34~36番目の3つ組の配列がコドンと対合するアンチコドン(注3)である。
注2)コドン
核酸(DNAとRNA)の塩基配列が、タンパク質を構成するアミノ酸配列へと生体内で翻訳されるときの、各アミノ酸に対応する3つ組(トリプレット)の塩基配列のこと。RNAの場合、アデニン(A)、グアニン(G)、ウラシル(U)、シトシン(C)の4塩基がトリプレットになるため、4×4×4=64通り存在する。64種類のコドンが、20種類のアミノ酸と翻訳終結の暗号を指定している。この関係は、遺伝暗号、遺伝コード(genetic code)等と呼ばれる。ここで登場するAUAコドンはアミノ酸のイソロイシンを指定するコドンである。
注3)アンチコドン
遺伝暗号をアミノ酸へと変換するため、tRNAの34~36番目に備わった3つ組の配列で、mRNA上のコドンと対合する。アンチコドン1字目(34番目)にしばしば修飾塩基が見られ、アグマチジン(図1)もこの位置にある。
注4)互変異性化
水素イオンが転移することで、その化学構造が変化すること。この場合は、シトシン環の4位のアミノ基の水素イオンが3位に転移し(図1)、一般的にはエナミン型からイミン型への互変異性化と捉えることができる。そのため塩基対合の様式が変化する。
注5)比較ゲノム
ゲノム配列が決定された複数の生物種に、共通に存在する遺伝子を抽出することで、目的の遺伝子を探索する手法。ここでは、アグマチジン修飾を持っている一群のアーキアに共通の遺伝子を絞り込んだ結果、最終的にTiaSを発見したということ。