機能不明のRNAを解明する新たな糸口を発見 ―RNAの安定性が機能と密接な関係にあることを解明―研究成果

機能不明のRNAを解明する新たな糸口を発見 |
平成24年3月27日
東京大学アイソトープ総合センター
1.発表者:
秋光信佳(東京大学アイソトープ総合センター 准教授)
井尻憲一(東京大学アイソトープ総合センター 教授)
鈴木 穣(東京大学大学院新領域創成科学研究科メディカルゲノム専攻 准教授)
谷 英典(東京大学アイソトープ総合センター 日本学術振興会特別研究員)
2.発表のポイント:
◆ヒト細胞中のRNAの安定性を網羅的に測定する手法を開発しRNAの安定性が機能と密接な関係にあることを明らかにした
◆これまで機能不明であった多数のノンコーディングRNAの機能を解明する道が開いた
◆RNAを標的とした疾病の原因解明や創薬開発への新しい応用研究につながる
3.発表概要:
東京大学アイソトープ総合センター 秋光信佳准教授、井尻憲一教授、谷 英典 日本学術振興会特別研究員、大学院新領域創成科学研究科メディカルゲノム専攻 鈴木 穣准教授らの研究グループは、ヒト細胞中のRNA (Ribonucleic Acidの略で、転写反応によってDNAを鋳型として合成される生体分子群)(注1)の安定性を網羅的に測定する方法を開発することで、RNAの安定性が機能と密接な関係があることを発見し、RNAの安定性を指標として機能不明なノンコーディングRNA(注2)の性質を定義できることを明らかにしました。この発見をもとに、これまで手がつけられていなかった多数のノンコーディングRNAを標的とした様々な疾病の原因解明や、これに基づく診断・創薬への応用に向けた研究が進展することが期待されます。
4.発表内容:
近年のゲノム科学の進展により、ヒトやマウスなどの哺乳動物のゲノムDNAから、機能不明のRNA群が大量に転写されていることがわかり、世界中から注目を集めています。これまでの遺伝子発現の概念では、DNAの塩基配列情報がメッセンジャーRNA(mRNA)に転写され、それがタンパク質のアミノ酸配列情報に翻訳されると定義されていました。しかしながら、新しく見つかったRNA群は、タンパク質のアミノ酸配列情報をコードせず、それ自身が独自の機能をもつのではないかと考えられています。これらはノンコーディングRNA(ncRNA)と呼ばれています。ゲノムから転写されるncRNAの割合は生物の複雑さに応じて増加し、ヒトでは転写産物の90%以上がncRNAによって占められることがわかってきており、ncRNAは細胞内で重要な調節機能を担っていると考えられていますが、その機能の大部分が解明されておりません。こうした現状から、ncRNAの機能解明によって、診断・創薬などの技術開発に新しい視点が現れるのではないかと期待が寄せられています。
これらのncRNAの中でも、マイクロRNA(micro RNA)などの数十塩基の長さを持つ短塩基長ncRNAの機能の理解は深まってきていますが、数百塩基以上の長さを持つ「長塩基長ncRNA」については、その機能のほとんどが未解明のままであり、機能未知の長塩基長ncRNAの機能を推定できるような革新的な指標が望まれていました。そこで我々は、 RNAの細胞内における安定性に着目しました。細胞の生存に必須なハウスキーピングタンパク質をコードするmRNA群は、細胞内で常に安定な状態で存在していますが、複雑な生理メカニズムを制御するような調節性タンパク質をコードするmRNA群は、細胞内で比較的不安定な状態、すなわち、細胞内で分解されやすいものが多いことがわかっています。従って、タンパク質をコードしないncRNAにおいても、RNAの安定性がその機能と密接に関係しており、RNAの安定性を指標とすることで機能不明な長塩基長ncRNAの性質を定義できるのではないかと考えました。
まず我々は、細胞内のRNAの安定性をmRNAからncRNAまで網羅的に測定できる手法として、最先端の技術である次世代シークエンサー(注3)を用いたBRIC-seq法(5’-BRomo-uridine Immunoprecipitation Chase - deep sequencing analysis)を開発しました。BRIC-seq法では、まず細胞の培地に5'-bromo-uridine(BrU)を添加し、そのまま一定時間培養してBrUを細胞内のRNAに取り込ませた後、培地を新しいものに交換することで、細胞内のRNAをBrUで短時間標識します。次に培地を交換してから経時的に細胞を回収していきます。このとき、半減期の短いRNAほどBrUの標識されていない通常のウラシルに置き換わっていきます。一方、半減期の長いRNAはBrU標識されたまま残ります。そして、それぞれの細胞から抽出したtotal RNAを用いて、抗BrU抗体によりBrU標識されたRNAのみを回収します。そして、次世代シークエンサーを用いてRNAの経時的な減少を測定し分解速度を求めます。BRIC-seq法を用いてヒト細胞中の11、052個のmRNA、1、418個のncRNAの安定性を測定しました(図1)。その結果、mRNAでは上記で予想された結果が得られ、さらに、ncRNAにおいては、細胞内で安定な状態で存在するRNA群(半減期4時間以上)にはハウスキーピング様の機能を有するncRNA(transfer RNAやsnoRNAなど)が多く含まれており、一方、不安定なRNA群(半減期4時間未満)にはHOTAIRやCDやCDKN2B-AS1などの調節性ncRNAが有意に濃縮されていることを発見しました。我々はこの不安定な分解されやすいncRNA群を、新しいカテゴリーとして「SLiTs(Short Lived non-coding Transcripts)」と名づけました。さらに、SLiTsに対して詳細な解析を進めた結果、細胞増殖に重要な役割を担う長塩基長ncRNAや、外部刺激により安定性が変化する長塩基長ncRNAを多数発見しました。以上の結果からRNAの安定性が長塩基長ncRNAの機能を定義する上で重要な指標となっており、その安定性によりこれまで機能不明であった長塩基長ncRNAの性質を定義できると考えられます。
本成果は、これまで手がつけられにくかった長塩基長ncRNAの理解を大きく前進させるものであり、その理解に基づく様々な疾病の原因解明や、診断・創薬へなどの応用研究の基盤となることが期待されます。
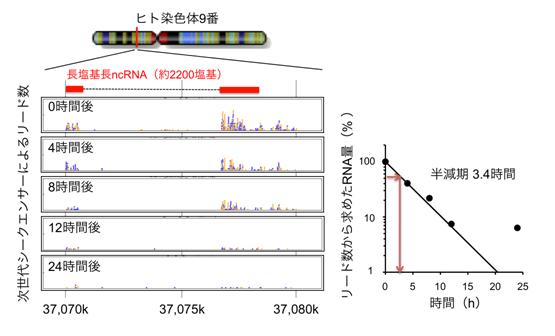
図1 BRIC-seq法の解析例として、ヒト染色体9番に存在する長塩基長ncRNAの安定性を測定した結果を示した。(図左)時間を経るごとに次世代シークエンサーにより読み取られる塩基配列のリード数が減少していく様子がみられ、RNAが分解されていくスピードがわかる。(図右)左のデータをグラフ化したもの。このようなデータがすべてのRNAに対して網羅的に解析できる。
5.発表雑誌:
雑誌名:「Genome Research」(オンライン版:2012年2月27日(米国東部時間))
論文タイトル:Genome-wide determination of RNA stability reveals hundreds of short-lived non-coding transcripts in mammals
著者:谷 英典、水谷 玲菜、Kazi Abdus Salam、田埜 慶子、井尻 憲一、若松 愛、磯貝 隆夫、鈴木 穣、秋光 信佳
DOI番号:10.1101/gr.130559.111
アブストラクトURL:http://genome.cshlp.org/content/early/2012/02/27/gr.130559.111.abstract
6.問い合わせ先:
東京大学アイソトープ総合センター 准教授 秋光信佳(あきみつ のぶよし)
7.用語解説:
(注1)RNA
リボ核酸。リボヌクレオチドがホスホジエステル結合で鎖状になった核酸。RNAのヌクレオチドはリボース、リン酸、塩基から構成されます。塩基とは、DNA及びRNAを構成する成分である複素環式化合物のことであり、アデニン(A)、グアニン(G)およびシトシン(C)はDNAにもRNAも含まれ、チミン(T)はDNAにのみ、ウラシル(U)はRNAにのみ存在します。
(注2)ノンコーディングRNA
タンパク質のアミノ酸配列をコードしないRNAの総称。Non-coding RNAを略してncRNAと呼ばれています。ノンコーディングRNAはタンパク質に翻訳されることなく、RNA自身が働いていると考えられています。ノンコーディングRNAは、現在少なくとも数千種類発見されていますが、大多数の機能は明らかになっていません。
(注3)次世代シークエンサー
遺伝子やゲノムなどのDNAの塩基配列を解析する装置をシークエンサーと呼び、次世代シークエンサーは従来のシークエンサーとは異なり、数千万~数十億の塩基配列情報を一度に得ることができます。