細胞染色画像からタンパク質の共変動ネットワークを構築 -「細胞状態」を活写する新解析法の開発により創薬の加速に期待- 研究成果
掲載日:2021年7月14日
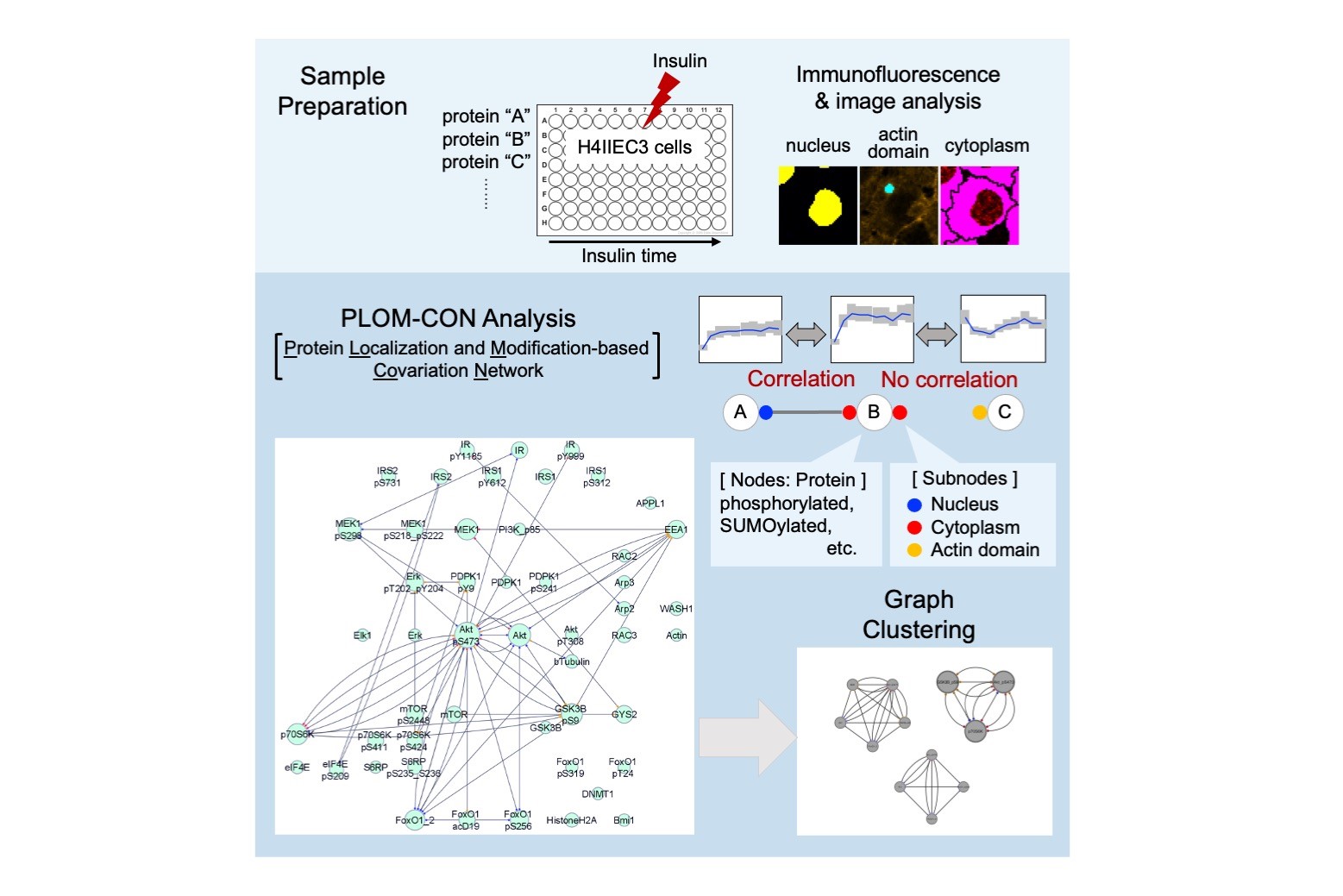
東京工業大学 科学技術創成研究院 細胞制御工学研究センターの加納ふみ准教授、中津大貴助教、國重莉奈特任助教、および東京大学 大学院総合文化研究科の村田昌之教授(研究当時。現:東京工業大学特任教授、東京大学名誉教授)、野口誉之助教(研究当時)、株式会社ニコンの米谷信彦研究員らの研究グループは、新しいタンパク質ネットワーク解析法である「PLOM-CON解析法」を開発し、従来の手法では困難だった細胞内に一過性に生じる2次元のタンパク質凝集体で、生化学的に単離・分析が困難なアクチンドメインの機能解明に成功した。アクチンドメインは糖尿病状態の肝細胞では形成されにくくなる研究結果もあり、この解析法が新しい創薬プラットフォームツールになることを実証した。
従来のタンパク質ネットワーク解析法は、生化学的手法で多数の細胞を集め破壊して得られるタンパク質の平均的な「量」の情報のみに基づいているが、タンパク質の「質・局在」情報の反映なくしては、創薬研究に重要な細胞のシグナル伝達を正しく理解できないことが課題であった。そこで、先端的な創薬・医療研究分野では、ヘテロさを特徴とする細胞集団や組織内の単一細胞が持つそれらの情報を考慮し、疾患や薬効が示す細胞状態をタンパク質のネットワークの変化によって知るための新しい解析技術が求められていた。
今回開発したPLOM-CON解析法では、細胞の蛍光抗体染色画像ビッグデータから得られる単一細胞レベルのタンパク質の量・質・局在情報を基に、タンパク質ごとの特徴量を算出し、それらの時間的相関の強弱を指標にして「共変動ネットワーク」として可視化できる。つまり、細胞へのシグナル入力に応答して得られる共変動ネットワークの変化や違いから、細胞状態の違いや変化を担うタンパク質群情報を知ることができる。
具体的に、ラット肝細胞内において、インスリン刺激によって一過性に形成される直径数mのアクチンドメインの機能を本解析法を用いて解析した結果、アクチンドメインがグリコーゲン合成やタンパク合成に関わる酵素群の集積とその制御を司る重要な場として機能することを実験的に明らかにした。
この「共変動ネットワーク」によって、肝細胞のみならず、様々な細胞のイメージング画像から得られる細胞の形態情報や、タンパク質を含む多様な生体分子の量や質的変化(多階層オミクスデータ)とを、相関解析的手法によって統合できると期待される。そのため、今回開発した新しい解析方法は、細胞を利用した基礎生命科学に留まらず、創薬や細胞医薬の新たな支援技術として活用できることの実証を連携企業とともに進めている。
研究成果は「iScience」で2021年7月13日(現地時間)に公開された。
従来のタンパク質ネットワーク解析法は、生化学的手法で多数の細胞を集め破壊して得られるタンパク質の平均的な「量」の情報のみに基づいているが、タンパク質の「質・局在」情報の反映なくしては、創薬研究に重要な細胞のシグナル伝達を正しく理解できないことが課題であった。そこで、先端的な創薬・医療研究分野では、ヘテロさを特徴とする細胞集団や組織内の単一細胞が持つそれらの情報を考慮し、疾患や薬効が示す細胞状態をタンパク質のネットワークの変化によって知るための新しい解析技術が求められていた。
今回開発したPLOM-CON解析法では、細胞の蛍光抗体染色画像ビッグデータから得られる単一細胞レベルのタンパク質の量・質・局在情報を基に、タンパク質ごとの特徴量を算出し、それらの時間的相関の強弱を指標にして「共変動ネットワーク」として可視化できる。つまり、細胞へのシグナル入力に応答して得られる共変動ネットワークの変化や違いから、細胞状態の違いや変化を担うタンパク質群情報を知ることができる。
具体的に、ラット肝細胞内において、インスリン刺激によって一過性に形成される直径数mのアクチンドメインの機能を本解析法を用いて解析した結果、アクチンドメインがグリコーゲン合成やタンパク合成に関わる酵素群の集積とその制御を司る重要な場として機能することを実験的に明らかにした。
この「共変動ネットワーク」によって、肝細胞のみならず、様々な細胞のイメージング画像から得られる細胞の形態情報や、タンパク質を含む多様な生体分子の量や質的変化(多階層オミクスデータ)とを、相関解析的手法によって統合できると期待される。そのため、今回開発した新しい解析方法は、細胞を利用した基礎生命科学に留まらず、創薬や細胞医薬の新たな支援技術として活用できることの実証を連携企業とともに進めている。
研究成果は「iScience」で2021年7月13日(現地時間)に公開された。
論文情報
Yoshiyuki Noguchi, Fumi Kano, Nobuhiko Maiya, Chisako Iwamoto, Shoko Yamasaki, Yosuke Otsubo, Daiki Nakatsu, Rina Kunishige, and Masayuki Murata, "Microscopic image-based covariation network analysis for actin scaffold-modified insulin signaling," iScience: 2021年7月13日, doi:10.1016/j.isci.2021.102724.
論文へのリンク (掲載誌 )
)