ライブセル・オミクスの実現へ 細胞内の網羅的な遺伝子発現量の非破壊・迅速測定を実現するラマン計測

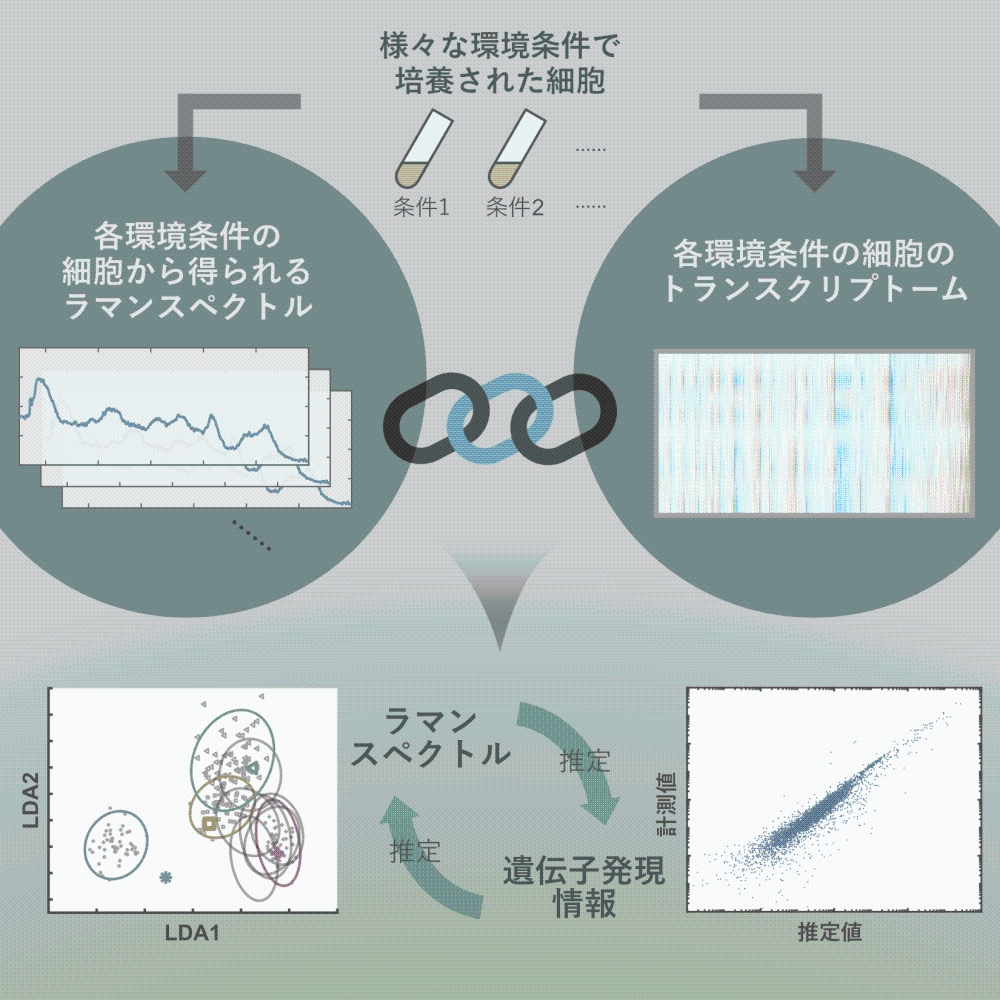
東京大学大学院総合文化研究科の若本祐一准教授と小林鉱石大学院生らの研究グループは、細胞にレーザー光を照射した際に得られるラマンスペクトルと、同培養環境に置かれた細胞内のトランスクリプトーム(網羅的な遺伝子発現量)のあいだに密接な対応関係があることを明らかにしました。さらにこの対応関係を利用することで、わずか数秒のレーザー光照射で取得できるラマンスペクトルから、細胞を破壊することなしに、細胞内の数千種類におよぶ網羅的な遺伝子発現量を推定できることを示しました。
細胞内には膨大な種類の遺伝子が存在し、その多数の遺伝子の発現状態によって細胞の状態が決定されると考えられています。これまで、トランスクリプトームやプロテオームなどの網羅的な遺伝子発現状態の計測手法では、必ず細胞を破砕または溶解し、細胞内のRNAやタンパク質を取り出す必要がありました。一方、細胞を破壊せずにその内部の網羅的な分子情報を得る手法として、ラマン分光法が注目されています。ラマン分光法とは、ある波長のレーザーを分子に当てた際、分子振動に依存してエネルギーシフトが生じた分子固有のラマン散乱光が生じることを利用し、このラマン散乱光のスペクトルを、いわば分子の指紋として標的分子を同定する手法です。しかし細胞は膨大な種類の分子から成り立っています。したがって、細胞から得られるラマンスペクトルは、多数の分子それぞれから生じるラマンスペクトルの重ね合わせとなります。この重ね合わさった極めて複雑なスペクトルから、細胞内部の大多数の分子の量を同定することは、スペクトルを分解するという従来の一般的な方法では不可能ではないかと考えられてきました。
今回、本研究グループは、ラマンスペクトルを直接分解するのではなく、細胞のトランスクリプトームとラマンスペクトルの対応を計算論的に見つけるという、新たなアプローチを採用しました。この研究ではまず、さまざまな環境条件に置かれた分裂酵母の細胞から得られるラマンスペクトルを主成分線形判別法と呼ばれる手法で次元圧縮することで、細胞が置かれていた環境条件に依存して、低次元空間内で細胞のラマンスペクトルが異なるクラスターを形成することを明らかにしました。さらにこの次元圧縮されたラマンスペクトルと遺伝子の発現を調べることのできるRNA-Seqによって取得されたトランスクリプトームとの対応関係を部分的最小二乗回帰で推定しました。得られた対応関係を元に、推定時に意図的に除外されたデータを使って、ラマンスペクトルからトランスクリプトームを推定すると、実際のトランスクリプトームのデータと高い精度で一致することを明らかにしました。統計テストにより、この高い精度の一致が偶然生じる確率は0.04%以下であることも示され、ラマンスペクトルとトランスクリプトームの対応関係はほぼ間違いないことが確認されています。また、同じ方法を大腸菌にも適用し、大腸菌においてもラマンスペクトルとトランスクリプトームのあいだに対応関係があり、ラマンスペクトルからトランスクリプトームを高い精度で推定できることが示されました。ラマンスペクトルからトランスクリプトームを予言できるためには、数千種類ある遺伝子の発現変動が、低次元空間内に制限されている必要があります。したがって今回の研究結果は、細胞内の遺伝子発現の変動が本質的には低次元であることも示唆しています。
ラマンスペクトルは、細胞へのわずか数秒のレーザー照射で得ることができます。また従来のトランスクリプトームやプロテオームなどのオミクス計測とは異なり、細胞を破壊することなしに、生細胞を対象に得ることができます。このラマン-トランスクリプトーム対応を利用したオミクス計測では、RNAやタンパク質を細胞から抽出し計測用に調製する作業も必要ありません。したがってこの対応を利用すれば、細胞のオミクス計測を、非破壊・迅速・安価に行える可能性があります。さらに、生細胞内の大規模な遺伝子発現の変化を、ラマンスペクトルを通して明らかにする「ライブセル・オミクス」の実現につながる可能性もあります。
「今回解析対象となった細胞内のトランスクリプトームが細胞の全質量に占める割合はわずか2%程度であり、RNAのラマンシグナルの強さも考慮すれば、細胞由来のラマンスペクトルが直接トランスクリプトームのシグナルを反映している可能性は非常に低いと思われます。にも関わらず、ラマンスペクトルとトランスクリプトームに対応が存在することは、細胞の分子構成の決定機構を考える上で大変興味深い結果です」と若本准教授は話します。「この研究結果は、生きた細胞からオミクス情報を取得できる道を拓くという応用上の重要性とともに、細胞内にある膨大な数の遺伝子発現の変化が比較的低次元の世界で理解できる可能性を示唆しており、細胞の動作原理を理解するうえで重要だと考えています」。
論文情報
Koseki J. Kobayashi-Kirschvink, Hidenori Nakaoka, Arisa Oda, Ken-ichiro F. Kamei, Kazuki Nosho, Hiroko Fukushima, Yu Kanesaki, Shunsuke Yajima, Haruhiko Masaki, Kunihiro Ohta, Yuichi Wakamoto, "Linear Regression Links Transcriptomic Data and Cellular Raman Spectra," Cell Systems: 2018年6月20日, doi:10.1016/j.cels.2018.05.015.
論文へのリンク (掲載誌 )
)