細胞が無駄なエネルギーを消費しないための遺伝子スイッチ停止機構の解明-ブドウ糖飢餓時におけるリボソーム遺伝子の発現抑制機構-研究成果

掲載日:2022年3月28日
発表者
平井 隼人(東京大学 大学院総合文化研究科 広域科学専攻 特任研究員)
太田 邦史(東京大学 大学院総合文化研究科 広域科学専攻 教授)
発表のポイント
- 栄養の飢餓によって、リボソーム遺伝子領域にヘテロクロマチン(注1)が強く形成され、遺伝子の発現スイッチがオフになる機構を明らかにしました。
- この機構が失われると、エネルギー源が乏しい中でリボソームRNA(注2)が過剰に産生され続け、細胞増殖や生存に大きな影響が生じることがわかりました。
- 今回発見した機構は、がんの発生や老化とも関係があると考えられ、がん治療や寿命の延長などに貢献する可能性があります。
発表概要
私たちの身体は約37兆個の細胞から構成されています。それぞれの細胞の核の中には生命の設計図であるDNAが格納されていて、この設計図を元に様々な種類のタンパク質が作られます。タンパク質が作られるためには、まずDNAからメッセンジャーRNA(mRNA)が転写され、その後、mRNAの情報をもとにタンパク質が合成されます。このタンパク質合成において中心的な役割を果たすのがリボソーム(注3)と呼ばれるRNA-タンパク質複合体です。細胞内では約6割から8割のエネルギーがリボソームの合成に費やされていることから、「タンパク質合成装置」であるリボソームを細胞内で維持するには多くの栄養(エネルギー)が必要となります。しかしながら、生物は常に栄養が豊富な環境で生育できるわけではありません。ときにはエネルギー源が不足した状況に陥ることもあります。その際に、豊富な栄養状況と同じようにリボソームが合成され続ければ、瞬く間に細胞内のエネルギーが枯渇することが予想されます。このようなエネルギー不足に陥るのを防ぐためには、エネルギー源の枯渇を感知してリボソームの合成を一旦停止する必要があると考えられます。本研究グループでは以前、分裂酵母細胞が栄養飢餓に直面すると、リボソーム合成に関連する遺伝子群の転写量が短時間で減少することを見出していました。しかしそのメカニズムは謎のままでした。
今回、本研究グループは、京都大学の竹俣直道助教と共同で、リボソーム遺伝子が複数連なっているDNA領域(rDNA)がブドウ糖飢餓によってヘテロクロマチン化され、リボソームRNAの転写が抑制されることやその仕組みを明らかにしました。また、飢餓時にヘテロクロマチンが形成されないと、リボソームRNAが過剰に転写され続け、細胞増殖の遅れや細胞死を引き起こすことがわかりました。
悪性腫瘍の原因となるがん細胞ではリボソームが過剰に産生され、生体内のエネルギーを横取りしながら活発に増殖し続けます。また、栄養飢餓により細胞寿命が延長される傾向があることが知られています。今回明らかになった機構は、がんの発生や老化とも関係があると考えられ、がん治療や健康寿命の延伸などに結びつく可能性があります。
発表内容
研究の背景
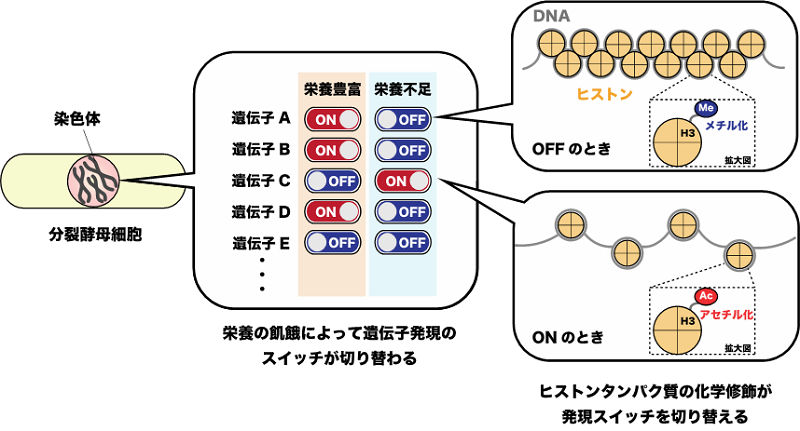
地球上に生息するすべての生物は、常に栄養が豊富な環境で生育できるわけではなく、栄養が不足した状況に直面するリスクを抱えています。そのため生物は長い年月をかけて、栄養の不足を切り抜けるための様々な術を獲得してきました。例えば、2016年にノーベル賞を受賞された大隅良典先生が発見したオートファジー(注4)もその1つです。また、微生物の中には栄養不足の環境下で胞子を形成し、栄養をほとんど消費しない休眠モードに入るものもいます。これらの現象は、いずれも遺伝子の発現スイッチが切り替わることで誘発されます。本研究グループは、分裂酵母細胞を栄養飢餓に直面させると多くの遺伝子で発現スイッチが切り替わることを見出していました(図1)。このスイッチの切り替えは、DNA自体あるいはDNAが巻き付くヒストンタンパク質がメチル化やアセチル化などの化学的な修飾を受けることで起こります(図1)。これまでに本研究グループは、エネルギー源であるブドウ糖の飢餓時に、ある種の遺伝子の発現スイッチがオンになる仕組みを明らかにしてきました。一方で飢餓によって遺伝子の発現スイッチがオフになる仕組みは、国内外でもあまり研究されておらず、大きな謎が残っていました。
研究内容
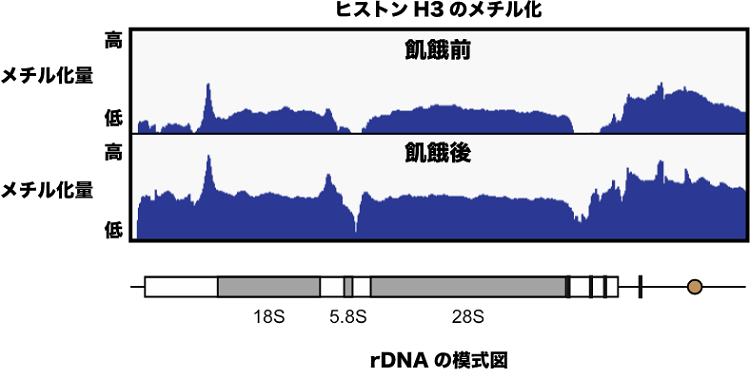
本研究グループの先行研究では、分裂酵母細胞が飢餓に直面するとリボソームに関連する遺伝子の多くが発現スイッチをオフにすることがわかっていました。リボソームは細胞内のエネルギーの多くを消費するため、飢餓時に発現が減少するのは理にかなっているといえます。そこで私たちは、リボソーム関連遺伝子の発現スイッチがオフになる仕組みの解明を目指しました。発現スイッチは、DNAがきつく巻き付いたヒストン同士が凝集した構造(ヘテロクロマチン)をとることでオフになります。これは4種類あるヒストンタンパク質の1つであるヒストンH3の9番目のアミノ酸残基(H3K9)がメチル化を受けることで達成されます(図1)。実際に、分裂酵母のゲノム全域を対象にH3K9のメチル化状態を調べたところ、リボソーム遺伝子が連なって存在するゲノム領域(rDNA)のヒストンH3が、ブドウ糖飢餓によって強くメチル化を受けることがわかりました(図2)。さらに、過去の知見をもとに、スイッチのオン・オフを司る因子としてAtf1(注5)とFACT(注6)と呼ばれる2つのタンパク質を同定しました。栄養が豊富なときはAtf1がrDNA上でFACTが集積するのを阻害する一方で、栄養が枯渇するとAtf1はrDNAから解離し、FACTが集積します。この2つのタンパク質の入れ替わりが、ヘテロクロマチンを形成して遺伝子の発現スイッチをオフにするトリガーであることがわかりました(図3)。この機能を欠損させると、飢餓状態でもリボソームRNAが過剰に産生され続け、細胞増殖の遅延や細胞死を引き起こすことも明らかになりました。
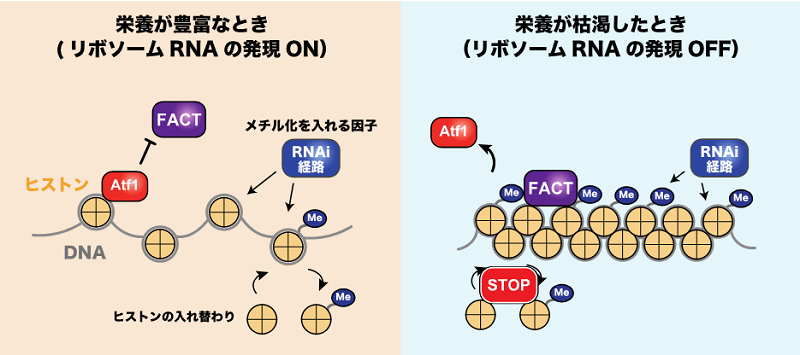
栄養が豊富なとき:Atf1タンパク質がヒストンシャペロンタンパク質FACTを阻害します。ヒストンH3はメチル化修飾を受けますが、ヒストンの入れ替わりが激しいためメチル化状態は低く維持されます。
栄養が枯渇したとき:Atf1が解離してFACTがrDNA上に集積します。ヒストンの入れ替わりが停止することでメチル化されたヒストンH3が増加します。
影響・波及効果
現代日本において、およそ4人に1人はがんにより亡くなっています。がん細胞ではリボソームが過剰に産生されており、生体内のエネルギー(ブドウ糖)を横取りしながら活発に増殖し続け、悪性腫瘍を形成します。したがって、がん細胞の過剰なリボソーム産生を抑制することができれば、治療につながると考えられます。また、リボソーム病(注7)と呼ばれるいくつかの疾患は、リボソーム遺伝子に傷が入ることで発症します。しかし発症機序には不明な点が多く、遺伝子発現のオン・オフの異常が影響を及ぼすのかはっきりとしたことはわかっていません。今回、本研究グループは発現スイッチを司るタンパク質を同定することに成功しました。高等生物でも保存された機構が発見されれば、がんやリボソーム病の治療薬の開発などにも進展することが期待されます。また、近年では、適度な栄養制限が寿命の延伸につながる可能性が指摘されています。さらに、列をなして多数存在するリボソーム遺伝子群の安定性は細胞の老化と密接に関係していることが報告されています。本研究をさらに発展させることで、栄養制限と長寿の関係について解き明かすことができるかもしれません。
本研究は、JST CREST研究費(JPMJCR18S3)の助成により支援されました。
用語解説
(注1)ヘテロクロマチン
遺伝情報を担うDNAを核内にコンパクトに収納するために、DNAはヒストンに巻き付いてヌクレオソームを形成し、さらにヌクレオソームが規則正しく折りたたまれることでクロマチン繊維を形成します。その中でも高度に凝集し転写が抑制された状態をヘテロクロマチンと呼びます。
(注2)リボソームRNA
細胞内のタンパク質を合成するリボソームはリボソームRNAとタンパク質から構成されています。リボソームRNAの転写量は全転写量の約6割から8割を占めているといわれており、細胞内のエネルギー資源の多くを利用します。分裂酵母では3番染色体の末端にリボソーム遺伝子が列をなして多数存在する領域(rDNA)があり、そこから転写されます。
(注3)リボソーム
ゲノムDNAから転写されたmRNAの情報をタンパク質へと変換する装置。活発に細胞分裂を繰り返すがん細胞ではリボソームが過剰に産生されていることや、リボソーム関連遺伝子の変異が疾患を引き起こす原因となることが報告されています。
(注4)オートファジー
細胞が栄養飢餓に陥ったときに細胞内のタンパク質を消化することで栄養源を確保する仕組み。
(注5)Atf1タンパク質
細胞が環境変化によるストレス刺激を受けると、ストレス応答性のMAPキナーゼ経路によってAtf1タンパク質がリン酸化を受けます。リン酸化されたAtf1はストレス応答性遺伝子の上流に結合することで遺伝子発現のスイッチを切り替えます。Atf1は高等生物にも保存されたタンパク質で、酵母と同様に環境ストレスに適応するための重要な機能をもちます。
(注6)FACTタンパク質
DNAの複製やDNAからmRNAが転写される際には、ヒストンとDNAの結合が一時的に緩まる必要があります。また、それらのイベントが終わると再びヒストンとDNAの結合が強まらなければなりません。このような再編成をヒストンシャペロンと呼ばれる種類のタンパク質が介助します。FACTタンパク質もその一種で酵母から高等生物まで広く保存されています。
(注7)リボソーム病
ヒトの疾患のいくつかは、リボソームに関連する遺伝子に傷が入ることで発症することがわかっています。このような疾患にはダイアモンド・ブラックファン貧血や先天性角化不全症、T細胞急性リンパ性白血病などが含まれます。
論文情報
Hayato Hirai, Naomichi Takemata, Miki Tamura, Kunihiro Ohta*, "Facultative heterochromatin formation in rDNA is essential for cell survival during nutritional starvation," Nucleic Acids Research: 2022年3月28日, doi:10.1093/nar/gkac175.
論文へのリンク (掲載誌 )
)